Tumor Mutational Burden (TMB)
;腫瘍遺伝子変異量
TMBは腫瘍遺伝子中の後天的に獲得された体細胞変異の総量であり、ネオアンチゲンの代替バイオマーカー(surrogate biomarker)となる可能性があります。がんの種類によって異なるものの、TMBによりI-O療法への反応性を予測できる可能性があります。また、TMBは次世代シーケンサー(NGS)を用いて測定することができます。
がんバイオマーカーとしての妥当性
- TMBは、腫瘍ゲノムにおける(後天的に獲得された)体細胞突然変異の総量として定義されます1,2。
- 腫瘍では、遺伝子変異量が高値であることと、ネオアンチゲンの予測数の増加は相関します3。
- 高いTMBは、腫瘍微小環境中の細胞傷害性T細胞
 の浸潤の程度と相関することが示されており、ネオアンチゲンを代替指標(surrogate
marker)として用いることを支持する知見となります4-6。
の浸潤の程度と相関することが示されており、ネオアンチゲンを代替指標(surrogate
marker)として用いることを支持する知見となります4-6。 - 腫瘍特異的なネオアンチゲンの存在量の増加により、がん細胞の免疫原性は強まり、腫瘍に浸潤する免疫細胞の数が増加します3,7。
- dMMRや環境中の変異原(タバコの煙や紫外線など)への曝露のような、明確な機序に基づくDNA変異によっても、TMBが高くなる可能性があります8,9。
予測的なI-OバイオマーカーとしてTMBの潜在的な有用性を検討する研究が現在進行中です。
それぞれのがんにおける頻度8
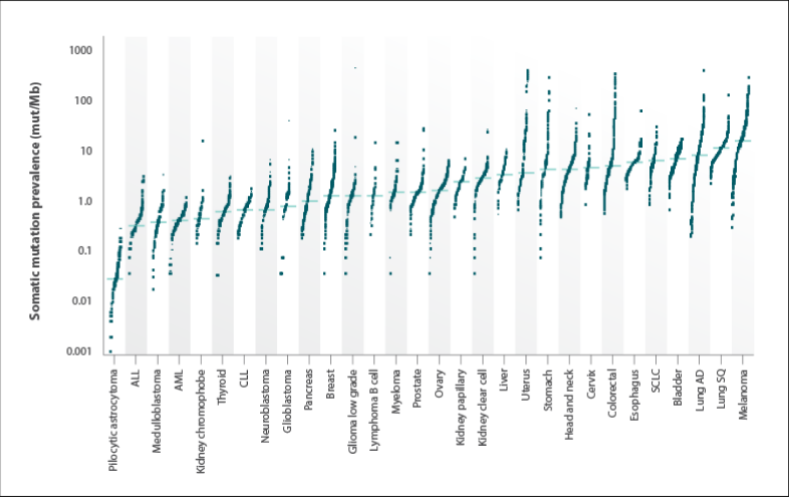
- 遺伝子変異の数は、腫瘍ごとに、そしてがんの種類によっても異なり、時間の経過(疾患の経過)とともに変化することもあります8-11。
- TMBが高い腫瘍は免疫系により認識され、標的とされる可能性が高いため、TMB検査により、抗腫瘍免疫応答の起こりやすさに関する情報が得られる可能性があります4-6。
- このように、TMBはがん細胞に対する免疫応答の可能性を予測できる新たなバイオマーカーであり、腫瘍の種類を問わず、個別化された治療に関する情報を得る上で役に立つ可能性があります3,12。
評価法
- TMBはNGSを用いて評価されます。NGSは、がん細胞のDNAを読み取り、参照ゲノムと比較して遺伝子変異を同定できる技術です13,14。
- TMBは次の3つの方法を用いて決定されます。全ゲノムの評価、全エクソームの評価、そして標的遺伝子パネルの使用の3つです1,15。
- 標的遺伝子パネルにより、あらかじめ特定された数百種類の遺伝子における変異と他の遺伝子変化を同時に検査することができ、TMBに加えて、従来のドライバー遺伝子変異を含む、重要な臨床的マーカーに関する情報が得られます13,16。
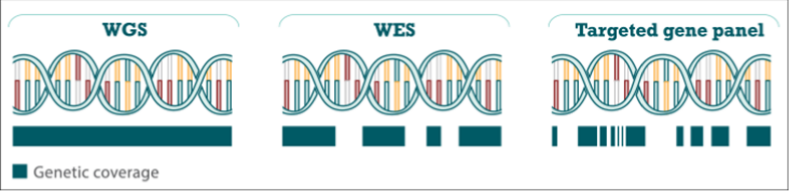
- より遺伝子数の多い標的遺伝子パネルを用いることで、腫瘍全体のTMBを高感度に定量できます1,17-19。
- TMBはがんの種類によって異なるため、TMBを高値と判定する閾値については、現在も検討されています1,9

TMB:I-O領域での研究
効果予測I-Oバイオマーカーとしての可能性について
REFERENCES–Tumor Mutational Burden (TMB);腫瘍遺伝子変異量
- Chalmers ZR, Connelly CF, Fabrizio D, et al. Analysis of 100,000 human cancer genomes reveals the landscape of tumor mutational burden. Genome Med. 2017;9(1):34. doi:10.1186/s13073-017-0424-2.
- Stratton MR, Campbell PJ, Futreal PA. The cancer genome. Nature. 2009;458(7239):719-724.
- Rooney MS, Shukla SA, Wu CJ, Getz G, Hacohen N. Molecular and genetic properties of tumors associated with local immune cytolytic activity. Cell. 2015;160(1-2):48-61.
- Chabanon RM, Pedrero M, Lefebvre C, Marabelle A, Soria JC, Postel-Vinay S. Mutational landscape and sensitivity to immune checkpoint blockers. Clin Cancer Res. 2016;22(17):4309-4321.
- Kim JM, Chen DS. Immune escape to PD-L1/PD-1 blockade: seven steps to success (or failure). Ann Oncol. 2016;27(8):1492-1504.
- Giannakis M, Mu XJ, Shukla SA, et al. Genomic correlates of immune-cell infiltrates in colorectal carcinoma. Cell Rep. 2016;15(4):857-865.
- Brown SD, Warren RL, Gibb EA, et al. Neo-antigens predicted by tumor genome meta-analysis correlate with increased patient survival. Genome Res. 2014;24(5):743-750.
- Alexandrov LB, Nik-Zainal S, Wedge DC, et al. Signatures of mutational processes in human cancer. Nature. 2013;500:415-421.
- Rizvi NA, Hellmann MD, Snyder A, et al. Mutational landscape determines sensitivity to PD-1 blockade in non-small cell lung cancer. Science. 2015;348(6230):124-128.
- Johnson BE, Mazor T, Hong C, Barnes M, et al. Mutational analysis reveals the origin and therapy-driven evolution of recurrent glioma. Science. 2014;343(6167):189-93.
- Wang J, Cazzato E, Ladewig E, et al. Clonal evolution of glioblastoma under therapy. Nat Genet. 2016;48(7):768.
- Yuan J, Hegde PS, Clynes R, et al. Novel technologies and emerging biomarkers for personalized cancer immunotherapy. J Immunother Cancer. 2016;4:3. doi:10.1186/s40425-016-0107-3.
- Frampton GM, Fichtenholtz A, Otto GA, et al. Development and validation of a clinical cancer genomic profiling test based on massively parallel DNA sequencing. Nat Biotechnol. 2013;31(11):1023-1031.
- Meyerson M, Gabriel S, Getz G. Advances in understanding cancer genomes through second-generation sequencing. Nat Rev Genet. 2010;11(10):685-696.
- Ng SB, Turner EH, Robertson PD, et al. Targeted capture and massively parallel sequencing of twelve human exomes. Nature. 2009;461(7261):272-276.
- Behjati S, Tarpey PS. What is next generation sequencing? Arch Dis Child Educ Pract Ed. 2013;98(6):236-238.
- Drilon A, Wang L, Arcila ME, et al. Broad, hybrid capture-based next-generation sequencing identifies actionable genomic alterations in lung adenocarcinomas otherwise negative for such alterations by other genomic testing approaches. Clin Cancer Res. 2015;21(16):3631-3639.
- Garofalo A, Sholl L, Reardon B, et al. The impact of tumor profiling approaches and genomic data strategies for cancer precision medicine. Genome Med. 2016;8:79. doi:10.1186/s13073-016-0333-9.
- Roszik J, Haydu LE, Hess KR, et al. Novel algorithmic approach predicts tumor mutation load and correlates with immunotherapy clinical outcomes using a defined gene mutation set. BMC Med. 2016;14(1):168. doi:10.1186/s12916-016-0705-4.
















